视紫红质:结构、成分与光信号捕捉
视紫红质:光量子捕捉机制、分子结构与其在视觉转导中的信号放大效应深度研究报告
1. 引言:生物学中的光子计数器
视觉不仅是感知光线的物理过程,更是生物体与环境信息交互的最重要渠道。在脊椎动物的视网膜中,这一过程始于一个令人惊叹的分子事件:单个光子被视杆细胞(rod cells)中的光受体蛋白——视紫红质(Rhodopsin)所捕获。视紫红质不仅是 G 蛋白偶联受体(GPCR)超家族的原型成员,更是自然界进化出的最完美的“光子计数器”之一。其在极低光照强度下(scotopic vision)的运作能力,使得生物体能够在仅有少量光子入射的环境中构建出清晰的视觉图像。1
本报告旨在对视紫红质进行穷尽式的深度剖析。我们将超越教科书式的简单描述,深入到原子层面的化学组成、飞秒(femtosecond)尺度的量子动力学以及宏观层面的信号放大机制。通过整合 X 射线晶体学、超快光谱学、单分子力谱学及分子动力学模拟的最新研究成果,本报告将回答三个核心问题:视紫红质由什么构成?其精细结构如何支持其功能?以及,它如何在量子力学和热力学的双重限制下,实现对光信号的极致捕捉与放大?
分析显示,视紫红质的高效性并非源于单一因素,而是源于其“预应力”发色团结构、蛋白质静电环境对能级间隙的量子调控、以及下游酶促反应的级联放大效应的完美协同。
2. 视紫红质的化学与元素组成分析
从化学角度审视,视紫红质是一个复杂的超分子复合物。其功能实现依赖于特定的元素配比、共价修饰以及金属离子的辅助。
2.1 蛋白质骨架:视蛋白(Opsin)
视紫红质的蛋白质部分被称为视蛋白,在牛视紫红质中,它是一条由 348 个氨基酸残基组成的多肽链,分子量约为 40 kDa。2
- 元素组成:其基础骨架由碳(C)、氢(H)、氧(O)、氮(N)和硫(S)构成。
- 硫元素的特殊角色:硫元素在视紫红质中不仅是甲硫氨酸和半胱氨酸的组成部分,更承担了关键的结构功能。特别是位于跨膜螺旋 III(TM3)顶端的 Cys110 和胞外环 2(ECL2)上的 Cys187 之间形成的一对高度保守的二硫键(Disulfide Bond)。这一共价键将 ECL2 紧紧束缚在受体顶部,形成一个“盖子”结构,这对稳定视黄醛结合口袋、防止化学噪声以及维持受体整体的三级结构至关重要。3
2.2 核心发色团:11-顺式视黄醛(11-cis-retinal)
视紫红质的光敏特性完全源于其结合的有机辅基——11-顺式视黄醛。
- 化学本质:它是维生素 A 醛的同分异构体,源自膳食中的 -胡萝卜素。在化学结构上,它是一个多烯链分子,具有高度共轭的 电子系统。4
- 席夫碱连接(Schiff Base Linkage):视黄醛并非通过非共价作用力结合,而是通过其醛基与视蛋白第 296 位赖氨酸(Lys296,位于 TM7)的 -氨基发生缩合反应,形成共价的席夫碱键(C=N)。
- 质子化状态(Protonated Schiff Base, PSB):在暗态下,该席夫碱是质子化的,带有一个正电荷(-NH=CH-)。这种质子化状态是视紫红质光谱特性的关键。游离的视黄醛吸收峰在紫外区(约 380 nm),而通过 PSB 与蛋白环境的相互作用(视蛋白位移,Opsin Shift),其吸收峰红移至 500 nm,使其能够有效吸收透过大气层的可见光。5
2.3 功能性金属离子:锌与钙
除了有机成分,微量金属元素在视紫红质的结构稳定性和功能调节中扮演着被长期忽视但极关键的角色。
- 锌离子(Zn):单分子力谱(SMFS)和分子动力学模拟揭示,锌离子能显著增强视紫红质内部结构片段的机械稳定性。锌离子通过与特定的组氨酸残基(如 His100 和 His195)以及其它配体形成配位键,稳定了受体的胞外结构域。6 这解释了为何视网膜中锌含量极高,以及缺锌为何会导致夜盲症及视网膜变性。锌不仅是结构稳定剂,还是光转导效率的潜在调节因子。7
- 钙离子(Ca)与离子泵功能:虽然脊椎动物视紫红质主要作为信号受体,但在进化的广阔视野下,某些微生物视紫红质充当光驱动的离子泵(如氯离子泵 Halorhodopsin 或质子泵)。在脊椎动物中,钙离子更多通过恢复蛋白(Recoverin)等调节蛋白间接参与光转导的负反馈调节,但视紫红质本身的某些结构域仍保留了与二价阳离子结合的保守模体。8
2.4 水分子:结构组分与反应物
水分子在视紫红质中不仅是溶剂背景,更是其分子机器的必要零件。
- 结构水(Structural Waters):X 射线晶体学显示,在跨膜螺旋之间存在一系列位置固定的有序水分子。这些水分子参与构建了复杂的氢键网络,连接了席夫碱与抗衡离子(Glu113),以及其它关键残基(如 Asp83, Gly120 等)。9
- 水解反应参与者:在光活化周期的末端,席夫碱必须水解以释放全反式视黄醛。同位素标记(O-labeled HO)实验证实,参与这一水解反应的水分子来自体相溶剂(bulk solvent),而非蛋白内部的结构水。这暗示了光活化过程中,蛋白构象的开放允许了外部水分子的流入。10
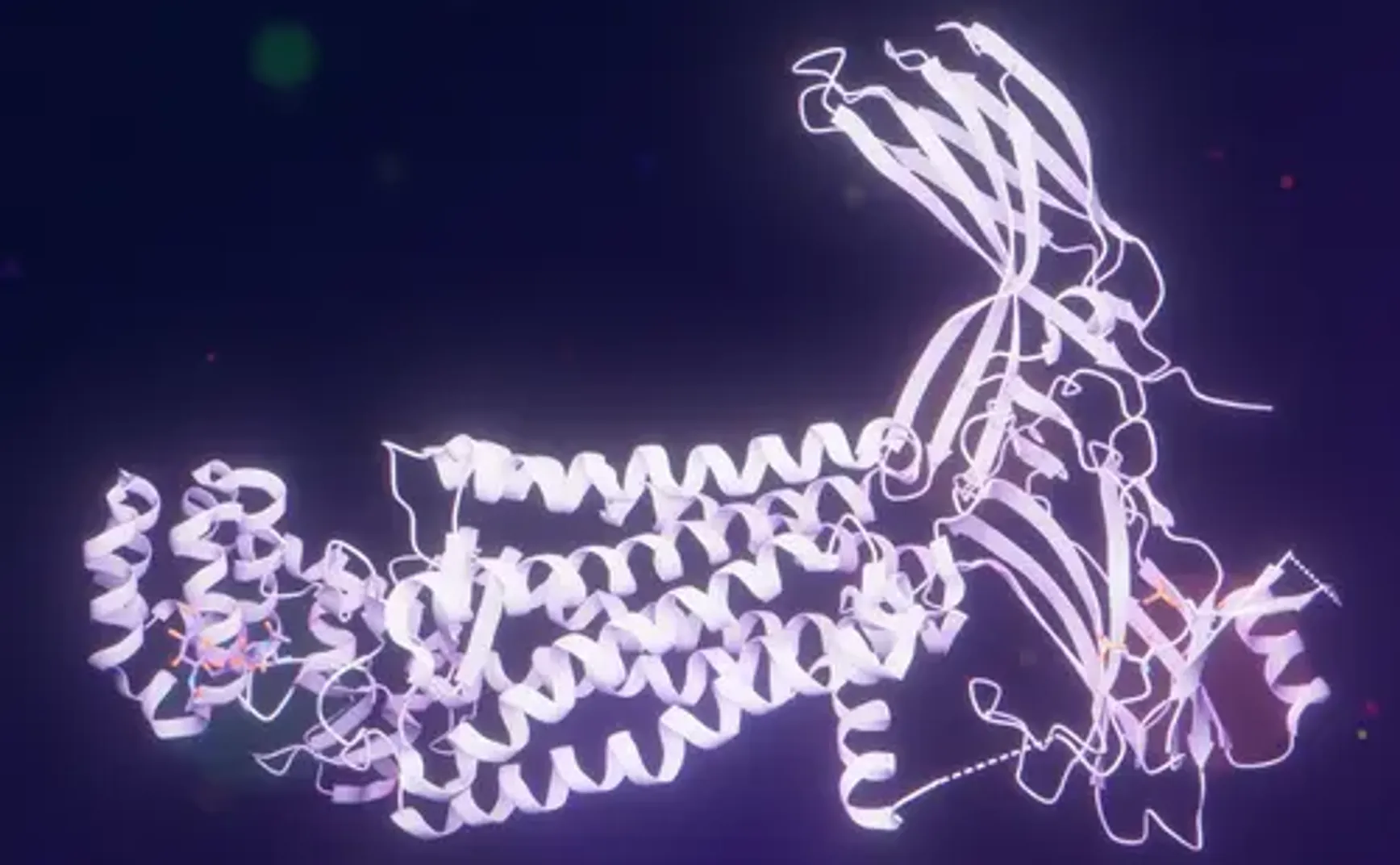
3. 分子结构与三维拓扑学特征
视紫红质的三维结构是其功能的物理载体。理解其结构不仅要看静态的晶体模型,更要理解其在膜环境中的动态组装。
3.1 七跨膜螺旋束(7TM Bundle)
视紫红质的跨膜结构域由 7 个 -螺旋(TM1-TM7)组成,它们以逆时针顺序排列(从胞外看),形成一个紧密的束状核心。
- 螺旋堆积:这些螺旋并非垂直穿过膜,而是以此特定的角度倾斜和扭曲,以适应内部的发色团。TM3 是结构的核心支柱,连接胞外和胞内结构域;TM7 则直接通过 Lys296 锚定发色团。11
- 晶体晶格中的二聚体:在晶体结构中,视紫红质倾向于形成反平行的二聚体。虽然这种特定的晶体接触可能不是生理状态下的主要形式,但它揭示了螺旋外侧疏水表面的性质,暗示了受体在视杆细胞膜盘上高密度排列(paracrystalline arrays)的可能性。12
3.2 视黄醛结合口袋与“离子锁”
11-顺式视黄醛深埋在由 TM3、TM4、TM5、TM6 和 TM7 围成的疏水空腔中,距离胞外表面约 15 Å。
- 空间位阻:结合口袋的形状与 11-顺式视黄醛的形状完美互补,但并非完全松弛。蛋白环境对发色团施加了扭曲力,迫使其 C11=C12 双键处于扭曲状态。这种“预应力”状态提高了其基态能量,降低了异构化所需的能垒,是实现超快光反应的关键结构基础。13
- 抗衡离子(Counterion Switch):带正电的质子化席夫碱是不稳定的,必须被负电荷中和。在暗态下,这一负电荷由 TM3 上的 Glu113 提供。Glu113 与 Lys296 之间通过直接的离子键或水介导的氢键相互作用,这被称为一级“离子锁”。光激活后,随着席夫碱位置的移动,抗衡离子会切换到 ECL2 上的 Glu181,这一“抗衡离子开关”机制对于光谱演化和受体激活至关重要。14
3.3 胞外结构域:稳定的“盾牌”
视紫红质的胞外部分包括 N 末端尾部和三个胞外环(ECL1-3)。
- N 末端糖基化:N 末端的 Asn2 和 Asn15 位点是糖基化的(N-glycosylation)。这些寡糖链在受体的折叠、细胞内运输以及膜盘的堆叠排列中起重要作用。15
- ECL2 的 -发卡结构:ECL2 不仅通过二硫键固定,还折叠成两股反平行的 -折叠片,深入受体核心上方。这个结构像盾牌一样阻挡了胞外亲水分子进入疏水核心,保护了对水敏感的席夫碱键免受自发水解,从而降低了系统的“化学噪声”。16
3.4 胞内结构域:信号传导界面
胞内部分包括三个胞内环(ICL1-3)和 C 末端尾部。
- 螺旋 8(Helix 8):在 TM7 之后,多肽链形成一个两亲性的短螺旋(H8),平行于膜表面。它被棕榈酰化(Palmitoylation)修饰,锚定在膜上。H8 是 G 蛋白识别和结合的关键部位。17
- 动态激活界面:在光激活过程中,TM6 会发生显著的向外刚体运动(outward tilt),打开原本封闭的胞内空腔,暴露出 G 蛋白结合位点。这一构象变化打破了胞内的另一组离子锁(Glu134-Arg135),标志着受体从静息态向活性态的转变。18
4. 高效捕捉光信号的量子动力学机制
用户提问的核心在于“为何能够高效捕捉光信号”。视紫红质的量子效率(Quantum Yield)约为 0.67,这意味着每吸收 3 个光子,就有 2 个能成功触发视觉信号。19 结合极低的热噪声,这种探测器接近了物理极限。
4.1 超快光异构化:飞秒化学的奇迹
光子吸收触发的首要事件是 11-顺式视黄醛绕 C11=C12 双键异构化为全反式构象。
- 时间尺度:这一过程在 200 飞秒(200 fs)内完成。20 作为对比,原子核的振动周期通常在 10-100 飞秒量级。这意味着反应快到分子甚至来不及进行热弛豫,能量被高效地用于驱动异构化,而非耗散为热量。
- 相干核运动(Coherent Nuclear Motion):与传统的随机热运动不同,光激发在势能面上产生了一个相干的波包(Wavepacket)。这个波包沿着反应坐标“弹道式”地运动,就像滑雪者从陡坡直冲而下。研究表明,这种相干性是由三个关键振动模式耦合驱动的:
- 模式:C11=C12 键的扭转。
- / 模式:氢原子的面外摇摆(HOOP)。
- BLA 模式:骨架键长交替。21
4.2 圆锥交叉(Conical Intersection, CI):量子漏斗
为何异构化如此之快且不可逆?答案在于势能面的拓扑结构。
- 无辐射跃迁通道:在反应路径上存在一个特殊的区域,激发态(S1)与基态(S0)的能量面相交,形成圆锥交叉。这就像是一个“漏斗”。当激发的波包到达这里时,它能以极高的概率瞬间穿越到基态的全反式一侧,而无需跨越热力学能垒。22
- 相位匹配:最新的量子-经典混合模拟显示,反应的成功率取决于波包到达 CI 时各振动模式的相位关系。特别是当氢原子摇摆速度()为正时,分子更有可能成功穿越 CI 成为产物。视蛋白的静电环境似乎进化为优化这一相位关系,从而最大化量子产率。23
4.3 极低的热噪声:信噪比的关键
高效捕捉信号不仅要求对光敏感,还要求对暗噪声免疫。
- 高能垒保护:在没有光的情况下,11-顺式视黄醛要想热异构化为全反式,必须克服巨大的能垒(约 45 kcal/mol)。这使得视紫红质在体温下的自发异构化半衰期长达 470 年。24
- 进化权衡:理论计算表明,通过改变蛋白静电场可以进一步提高量子产率至 0.7 以上,但这会同时降低基态能垒,导致热噪声指数级增加。现有的 0.67 效率与 470 年半衰期,是自然选择在“高灵敏度”与“低误报率”之间达成的最佳平衡。25
5. 光转导级联:信号的生化放大机制
捕获光子只是第一步。单个视紫红质分子的构象变化如何引发宏观的神经电信号?这依赖于多级酶促反应的信号放大(Amplification)。
5.1 光活化中间体序列
异构化后,视紫红质经历一系列热弛豫中间体,每一个都对应特定的构象和光谱特征。26
- Photorhodopsin (吸收峰 ~570 nm): 飞秒级产物,高度扭曲。
- Bathorhodopsin (吸收峰 ~543 nm): 皮秒级,能量储存态,拥有高基态焓。
- Lumirhodopsin (吸收峰 ~497 nm): 纳秒级。
- Metarhodopsin I (Meta I) (吸收峰 ~478 nm): 微秒级,席夫碱仍质子化。
- Metarhodopsin II (Meta II / R)* (吸收峰 ~380 nm): 毫秒级。此时席夫碱去质子化,TM6 向外倾斜,形成具有催化活性的 R* 态。
5.2 第一级放大:R* Transducin (Gt)
这是第一个生化放大步骤。R* 作为一个鸟苷酸交换因子(GEF),催化异三聚体 G 蛋白(Transducin, Gt)上的 GDP 被 GTP 置换。
- 放大倍数:一个 R* 在其活性寿命(约 50-100 ms)内,通过在膜表面的二维扩散,可以连续碰撞并激活 16-100 个转导蛋白分子(在冷血动物如青蛙中可达数百个)。27
- 速率:催化速率极高,达到 150-400 分子/秒。这种高效率得益于视盘膜上极高的蛋白密度(~25,000 个/),缩短了分子间的扩散距离。28
5.3 第二级放大:Gt PDE6 cGMP
被激活的 Gt-GTP 亚基与磷酸二酯酶(PDE6)的 抑制亚基结合,解除了 PDE6 的抑制。
- PDE6 的高效性:PDE6 是自然界效率最高的酶之一,其 接近扩散极限。
- 水解风暴:一个激活的 PDE6 分子每秒可水解数千个 cGMP 分子。综合来看,单个光子吸收最终导致 个 cGMP 分子的降解。29
5.4 信号输出:离子通道关闭
细胞质 cGMP 浓度的骤降导致环核苷酸门控通道(CNG 通道)关闭。
- 暗电流阻断:在暗态,CNG 通道开放,允许 Na 和 Ca 内流。光照使其关闭,引起膜电位超极化(从 -40 mV 降至 -70 mV)。
- 单光子响应:这种超极化足以中断谷氨酸的释放,产生约 1 pA 的电流变化,被下游的双极细胞检测到。这就是单光子视觉的生理基础。30
表 1:光转导级联中的放大参数概览
| 步骤 | 分子事件 | 放大倍数/速率 | 关键机制 |
|---|---|---|---|
| 光捕获 | 光子 R* | 1 : 1 (量子产率 ~0.67) | 异构化,圆锥交叉 |
| G 蛋白激活 | R* Gt* | 1 : ~20-100 | R* 作为催化剂,二维扩散 |
| PDE 激活 | Gt* PDE6* | 1 : 1 | 解除 亚基抑制 |
| cGMP 水解 | PDE6* cGMP | 1 : ~1000-2000 | 高 酶促反应 |
| 总增益 | 光子 cGMP | 1 : ~ | 多级级联 |
6. 信号终止与视觉循环:恢复灵敏度
为了保持时间分辨率并防止受体饱和,信号必须被迅速切断,视紫红质必须被再生。
6.1 快速关断(Quenching)
- 磷酸化:视紫红质激酶(GRK1)识别 R*,并在其 C 末端的 Ser/Thr 残基上添加磷酸基团。这降低了 R* 对 Gt 的亲和力。31
- 阻遏蛋白结合:视觉阻遏蛋白(Arrestin-1)高亲和力地结合磷酸化的 R*,彻底物理阻断其与 G 蛋白的接触。这一过程在光照后不到 100 毫秒内发生,确保了信号的瞬态性。32
6.2 视紫红质再生(Regeneration)
失活的 R* 最终会分解释放出全反式视黄醛和视蛋白。
- 席夫碱水解:这是视觉循环中最慢的一步,也是限速步骤。外部水分子进入口袋,水解席夫碱键。
- RPE 循环:全反式视黄醛被还原为全反式视黄醇(维生素 A),运送到视网膜色素上皮细胞(RPE)。在那里,RPE65 异构酶将其重新转化为 11-顺式视黄醛,再运回视杆细胞与视蛋白重组。这一复杂的穿梭过程称为“视觉循环”(Visual Cycle),是维持持续视觉能力的物质基础。33
7. 比较与临床视角
7.1 视杆与视锥的对比
虽然视锥细胞(负责色觉)也使用类似的视蛋白,但视杆视紫红质在稳定性上无与伦比。视锥色素的热噪声比视紫红质高出几个数量级,这也是为何我们无法用视锥细胞在极暗环境下看到颜色的原因——其信噪比不足以支持单光子检测。34
7.2 疾病与突变
视紫红质基因(RHO)的突变是导致视网膜色素变性(Retinitis Pigmentosa, RP)的主要原因之一。
- 结构失稳突变(如 P23H):导致蛋白错误折叠,滞留在内质网,引起细胞应激和死亡。
- 组成型激活突变(如 K296E):破坏了 Lys296 与 Glu113 的盐桥,导致受体在没有光的情况下持续激活,产生持续的“虚假光信号”,最终导致光感受器退化。35
8. 技术与仿生学启示
视紫红质的极致性能为现代技术提供了灵感。
- 单光子探测器:基于视紫红质原理设计的“FOCUS”探测器,模仿了其大吸收截面和纳米级门控机制,旨在实现室温下的红外单光子探测,克服了传统半导体探测器高噪声的缺陷。36
- 光遗传学:虽然光遗传学主要使用微生物视紫红质(Type I),但对动物视紫红质(Type II)结构机制的理解,正在推动开发具有更高灵敏度、更特异光谱响应的新一代光控工具。37
9. 结论
综上所述,视紫红质之所以能够高效捕捉光信号,并非偶然,而是自然界在数十亿年进化中对物理法则的极致利用。
- 化学上,它利用质子化席夫碱将量子的能量能级调整至可见光区,并利用共价键将发色团预应力固定。
- 物理上,它利用圆锥交叉的拓扑结构,在飞秒尺度上引导相干核运动,实现了近乎完美的量子效率,同时利用高能垒屏蔽热噪声。
- 生物上,它构建了一个高增益的信号放大级联,将微观的分子变形转化为宏观的神经信号。
视紫红质不仅是视觉的起点,更是量子生物学的杰作,是“结构决定功能”这一生物学核心法则最精妙的注脚。
未在文中直接引用但列出的参考资料
- Evolution of binding sites for Zinc and Calcium ions playing structural roles - ResearchGate, https://www.researchgate.net/publication/5841748_Evolution_of_binding_sites_for_Zinc_and_Calcium_ions_playing_structural_roles
- Functional role of internal water molecules in rhodopsin revealed by x-ray crystallography, https://www.pnas.org/doi/10.1073/pnas.082666399
- Transducin - Wikipedia, https://en.wikipedia.org/wiki/Transducin
- Current understanding of signal amplification in phototransduction - PMC - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC4160332/
- A Simplified Model of Activation and Deactivation of Human Rod Phototransduction—An Electroretinographic Study | IOVS, https://iovs.arvojournals.org/article.aspx?articleid=2792777
- Effect of Trace Metal Ions on the Conformational Stability of the Visual Photoreceptor Rhodopsin - PMC - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC10342768/
-
Rhodopsin - Wikipedia, https://en.wikipedia.org/wiki/Rhodopsin ↩︎
-
Rhodopsin - Wikipedia, https://en.wikipedia.org/wiki/Rhodopsin ↩︎
-
Rhodopsin - Proteopedia, life in 3D, https://proteopedia.org/wiki/index.php/Rhodopsin ↩︎
-
Microbial and Animal Rhodopsins: Structures, Functions, and Molecular Mechanisms | Chemical Reviews - ACS Publications, https://pubs.acs.org/doi/10.1021/cr4003769 ↩︎
-
Rhodopsin - Wikipedia, https://en.wikipedia.org/wiki/Rhodopsin ↩︎
-
Conserved rhodopsin intradiscal structural motifs mediate stabilization; effects of zinc - PMC, https://pmc.ncbi.nlm.nih.gov/articles/PMC2765562/ ↩︎
-
Stabilizing Effect of Zn2+ in Native Bovine Rhodopsin - PMC - PubMed Central, https://pmc.ncbi.nlm.nih.gov/articles/PMC2043472/ ↩︎
-
Rhodopsin - Proteopedia, life in 3D, https://proteopedia.org/wiki/index.php/Rhodopsin ↩︎
-
G Protein-Coupled Receptor Rhodopsin: A Prospectus - PMC, https://pmc.ncbi.nlm.nih.gov/articles/PMC1435697/ ↩︎
-
Role of Bulk Water in Hydrolysis of the Rhodopsin Chromophore - PMC - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC3099708/ ↩︎
-
Rhodopsin - Wikipedia, https://en.wikipedia.org/wiki/Rhodopsin ↩︎
-
G Protein-Coupled Receptor Rhodopsin: A Prospectus - PMC, https://pmc.ncbi.nlm.nih.gov/articles/PMC1435697/ ↩︎
-
Thermal Properties of Rhodopsin: INSIGHT INTO THE MOLECULAR MECHANISM OF DIM-LIGHT VISION - PMC - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC3149353/ ↩︎
-
Role of Bulk Water in Hydrolysis of the Rhodopsin Chromophore - PMC - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC3099708/ ↩︎
-
G Protein-Coupled Receptor Rhodopsin: A Prospectus - PMC, https://pmc.ncbi.nlm.nih.gov/articles/PMC1435697/ ↩︎
-
Rhodopsin - Proteopedia, life in 3D, https://proteopedia.org/wiki/index.php/Rhodopsin ↩︎
-
Rhodopsin - Proteopedia, life in 3D, https://proteopedia.org/wiki/index.php/Rhodopsin ↩︎
-
Deprotonation of retinal Schiff base and structural dynamics in the ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC12256820/ ↩︎
-
Quantum–classical simulations of rhodopsin reveal excited-state ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC8983576/ ↩︎
-
Quantum–classical simulations of rhodopsin reveal excited-state ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC8983576/ ↩︎
-
Quantum–classical simulations of rhodopsin reveal excited-state ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC8983576/ ↩︎
-
Quantum–classical simulations of rhodopsin reveal excited-state ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC8983576/ ↩︎
-
Quantum–classical simulations of rhodopsin reveal excited-state ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC8983576/ ↩︎
-
Biophysics and Quantum Limitation of Photoreceptive Processes - MDPI, https://www.mdpi.com/2673-8392/4/1/3 ↩︎
-
Quantum–classical simulations of rhodopsin reveal excited-state ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC8983576/ ↩︎
-
Rhodopsin - Wikipedia, https://en.wikipedia.org/wiki/Rhodopsin ↩︎
-
Full article: Current understanding of signal amplification in phototransduction, https://www.tandfonline.com/doi/full/10.4161/cl.29390 ↩︎
-
Phototransduction, Dark Adaptation, and Rhodopsin Regeneration The Proctor Lecture, https://iovs.arvojournals.org/article.aspx?articleid=2164197 ↩︎
-
Full article: Current understanding of signal amplification in phototransduction, https://www.tandfonline.com/doi/full/10.4161/cl.29390 ↩︎
-
Single-photon detection by rod cells of the retina - Center for Neural Science, https://www.cns.nyu.edu/csh/csh04/Articles/Rieke1998.pdf ↩︎
-
Deactivation Mechanisms of Rod Phototransduction: The Cogan ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC2845640/ ↩︎
-
11-cis and all-trans retinols can activate rod opsin: Rational design of the visual cycle - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC2561911/ ↩︎
-
11-cis and all-trans retinols can activate rod opsin: Rational design of the visual cycle - NIH, https://pmc.ncbi.nlm.nih.gov/articles/PMC2561911/ ↩︎
-
Deprotonation of retinal Schiff base and structural dynamics in the ..., https://pmc.ncbi.nlm.nih.gov/articles/PMC12256820/ ↩︎
-
Rhodopsin - Wikipedia, https://en.wikipedia.org/wiki/Rhodopsin ↩︎
-
A single photon detector inspired by the human eye - SPIE, https://www.spie.org/news/0988-a-single-photon-detector-inspired-by-the-human-eye ↩︎
-
Microbial and Animal Rhodopsins: Structures, Functions, and Molecular Mechanisms | Chemical Reviews - ACS Publications, https://pubs.acs.org/doi/10.1021/cr4003769 ↩︎